主要观点总结
本文主要介绍了使用自监督学习对未标注的病理切片进行组织形态癌症表型映射的研究,文章涵盖研究背景、方法、结果和讨论。该研究通过无监督深度学习方法识别组织形态表型簇,揭示了从良性到恶性组织的转化路径,对癌症的诊断和管理具有重要意义。
关键观点总结
关键观点1: 研究背景
癌症的诊断和管理依赖病理学家从显微图像中提取复杂信息,这个过程耗时且易受人为偏见影响。传统的监督式深度学习方法虽然有效,但受限于训练标注的成本和质量。该研究提出了一种自监督的方法——组织形态表型学习,无需标签,通过自动发现图像切片中的辨别性特征。
关键观点2: 研究方法
文章介绍了研究使用自监督学习对未标注的病理切片进行组织形态癌症表型映射的具体流程,包括数据预处理、自监督学习模型构建、组织形态表型簇识别等步骤。
关键观点3: 研究结果
研究通过自监督学习模型成功识别了肺腺癌中的不同组织形态学模式,建立了组织形态表型图谱(HP-Atlas),揭示了组织形态的转化路径。此外,该研究还发现了与临床结果相关的组织形态表型,为癌症的预后预测提供了有力工具。
关键观点4: 技术难点与解决方案
该研究在技术上虽然难度不大,但实验设计得很好,工作量巨大。从病理关联组织表型是一个好的思路,值得学习和模仿。通过自监督学习、社区检测、最近邻图(NNG)、UMAP等算法,解决了数据标注成本高、局部结构保留等问题。
关键观点5: 研究影响与展望
该研究为癌症的诊断和 management 提供了新的思路和方法,通过组织形态表型学习,提高了癌症诊断的准确性和效率。此外,该研究还展示了AI在医学领域的应用潜力,为未来的医学研究提供了新的方向。
正文
综上,病理和AI,还是可以持续加码的,但是有技术、有硬件、有资源,总结一句话,
要有钱
。

文章标题:
Mapping the landscape of
histomorphological cancer phenotypes
using self-supervised learning on
unannotated pathology slides
中文标题:
使用自监督学习对未标注的病理切片进行组织形态癌症表型映射
发表期刊:
《Nature Communications》
发表时间:
2023年8月11日
研究背景
(1)癌症的诊断和管理
依赖病理学家从显微图像中提取复杂信息,这个过程耗时且易受人为偏见影响。
(2)传统的监督式深度学习方法尽管有效,但受限于训练标注的成本和质量。
提出了一种自监督的方法——组织形态表型学习,
无需标签,通过自动发现图像切片中的辨别性特征
。
(3)
组织切片被分为形态上相似的簇,形成
组织形态表型图谱
(HP-Atlas),
揭示了从良性到恶性组织的转化路径,炎症和反应性表型。
这些集群特征可以利用正交方法识别,能够将组织学、分子和临床表型相联系。
(4)在肺癌研究中,表型簇与患者生存期、已识别的肿瘤类型和生长模式以及免疫表型的转录组学指标密切相关。
(5)概念介绍
这里涉及一些工科技术的概念,有利于理解该研究:
1)Leiden community detection:
Leiden 社区检测是一种用于揭示网络中社区结构的算法,旨在识别网络中的紧密连接子集(即社区)。该算法是根据 Louvain 方法的基础上发展而来的,具有更高的准确性和更有效的计算效率。
eiden 社区检测算法在生物信息学中的应用
主要通过解析复杂的生物网络结构,揭示功能相关的基因模块、蛋白质复合体、代谢途径、疾病相关基因群体和单细胞亚群,为研究和理解生物学过程提供了强大的工具和支持
。这些应用不仅有助于发现新的生物标志物和调控机制,还促进了个性化医疗和药物开发等领域的发展。
2)最近邻图(Nearest Neighbor Graph, NNG)
NNG算法是一种在机器学习和数据分析中广泛应用的
图构建方法
,用于表示
数据点之间的局部关系
。通过最近邻图,可以有效揭示
数据中的局部结构
,适用于聚类、降维、数据可视化和异常检测等多种任务。
3)UMAP(Uniform Manifold Approximation and Projection)
是一种用于
降维和数据可视化的算法
,基于流形学习理论,旨在
保留高维数据的全局和局部结构
。UMAP 首先通过最近邻搜索构建局部邻接图,并计算相似性,然后合并局部图形成全局图。该算法通过优化目标函数,使低维空间中的数据点布局尽可能反映高维空间中的相似性。与其他降维方法(如 PCA 和 t-SNE)相比,UMAP 具有效率高、结构保留良好及灵活性强的特点,广泛应用于数据可视化、
聚类分析、单细胞RNA测序等领域
。其简便的实现方式使其在处理各种类型的数据时成为一个受欢迎的选择。
探讨结果
1 HPL:通过自监督学习和社区检测进行组织形态表型学习
1.1 HPL方法概述
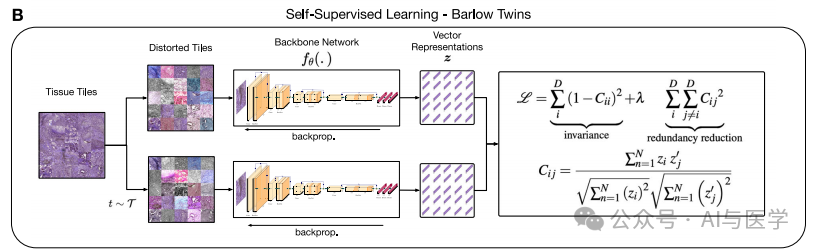
HPL(组织形态学表型发现)通过无监督的深度学习方法,在无需病理学家标注的情况下,自动识别全视野图像(WSI)中的不同组织形态学模式。

1.2 自监督学习
HPL通过自监督学习将
每个图块转化为128维的向量
表示,捕捉其形态特征,并确保对颜色和缩放失真不敏感。
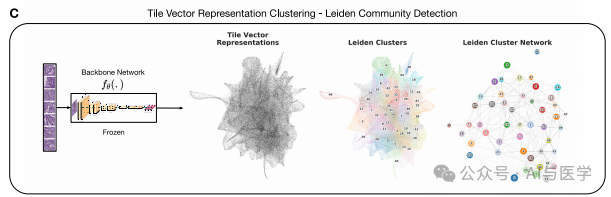
1.3 形态学表型簇(HPCs)
:
HPL通过最近邻图和Leiden社区检测算法,从图块中识别组织形态学表型簇(HPCs),并通过自监督方法选择合适的HPC数量。
1.4 HPL优势
HPL方法为组织模式
提供可视化和可解释性,支持与基因组和转录组等多组学特征的关联研究,并可量化空间异质性
。




















