正文
。
卵细胞中的的H3K4me3广泛存在,到受精后只存在于转录起始位点(Fig.1),在ESC发育不同时期基因组的水平上相对稳定。传统认为H3K4me3促进基因表达,但又不能直接作为基因转录激活的标记,并且激活机制尚不明晰。
b) H3K4me1、H3K4me2:
一般存在于基因间区域和远端的调控元件上,起到无差别激活的作用。H3K4me1由包含MLL3和MLL4的蛋白复合体催化(Fig.1),缺失MLL3的小鼠胚胎发育正常,但围产期致死;而MLL4突变体在胚胎期致死,说明MLL4在胚胎发育时期是必要的。H3K4me1的去甲基化酶LSD1突变后使得甲基化水平异常升高,ESCs和HSCs会不断增值,但不能继续向正确的方向分化。综上所述,H3K4me1并不会显著影响细胞性状维持,而是主要在细胞形态和身份转化过程中起作用
(
1
)
。
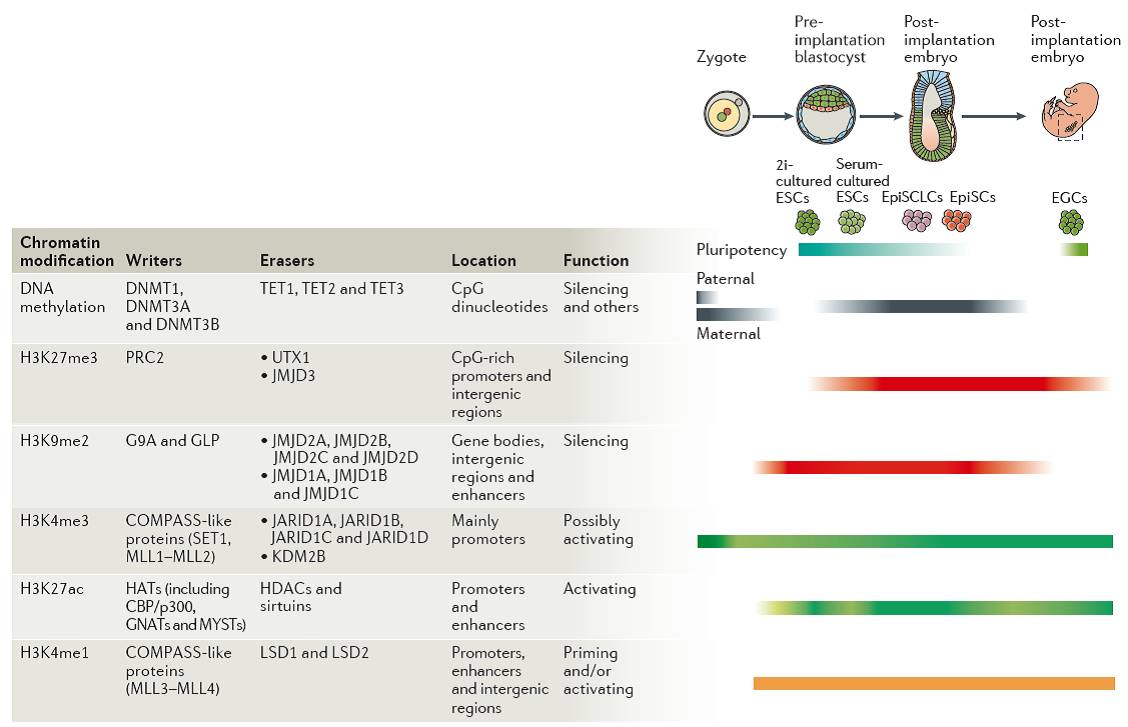
Fig.1 早期胚胎发育及多能干细胞分化过程中,不同表观遗传修饰及其调节蛋白的动态表达
c. H3K27乙酰化:
H3K27乙酰化主要存在于激活性的调控元件附近,比如基因的启动子、增强子等等。H3K27的乙酰化是由组蛋白乙酰转移酶(HATs)催化的,如CREB结合蛋白和p300(CBP/p300)等。H3K27ac经常与H3K4me1、H3K27me3共同作用,比如在鼠源的ESCs中,约有一半含有H3K4me1的增强子区域同时也有H3K27ac的修饰;并且大多数的H3K27ac标记的增强子区域含有H3K4me1的标记。大约70%包含H3K27ac标记的增强子都处于激活状态,能够在体内促进基因的转录
(
1-3
)
。
组蛋白去乙酰化酶HDAC1主要影响早期胚胎中组蛋白去乙酰化作用,Hdac1突变体中,胚胎的囊胚发育被延迟、并在着床后致死。缺失HDAC1的体外培养的ESCs全能性持续存在,但增殖能力减弱。
BET(Bromo- and extra- terminal)蛋白是组蛋白乙酰化的识别者,通过识别乙酰化、介导相应的反应通路。BET蛋白的敲除、功能缺失以及抑制剂处理,都会导致胚胎致死,再次佐证了H3K27ac影响ESC细胞自我更新和胚胎发育
(
3
)
。
2)抑制型的组蛋白修饰:
a. H3K27me3
H3K27me3修饰主要位于启动子的CpG岛区域、以及基因间区域,与基因沉默相关(Fig.1)。并且,H3K27me3一般处于DNA甲基化水平比较低甚至未被甲基化的区域,比如HOX基因簇(homeobox)。H3K27me3也起到维持雌性细胞被失活的那条X染色体转录抑制状态的作用。PRC2(Polycomb repressive complex 2)能够催化H3K27me3的形成,在ESCs的早期分裂和着床前发育过程中并没有功能,只有在原肠胚开始分化后,PRC2的功能缺失会导致胚胎死亡。总结而言,H3K27me3对转录的抑制功能在原初的胚胎干细胞中不是必要的;然而在后续分化时期、特别是在着床后发育过程中至关重要。另外,H3K27me3更多的是作为一个基因沉默的标志,比如在缺失PRC2组分的小鼠ESCs中,转录抑制往往发生在发育过程进行到一定阶段后,也就是说H3K27me3的标记是作为基因沉默的维持者工作,而不是导致其沉默的起始原因
(
2-3
)
。
b. H3K9me2
H3K9me2与基因沉默相关,在染色体上的分布经常以超大尺度存在,甚至可能超过4Mb,一般分布在基因本体和基因间区域(Fig.1)。催化H3K9me2的蛋白复合体中主要包含G9A(EHMT2)和GLP(EHMT1),缺陷的小鼠胚胎都会在E9.5时致死,但ESCs能够被持续培养,证明H3K9me2在胚胎发育过程中起作用,但在ESCs分化过程中尚存争议
(
3-4
)
。
1.2 DNA修饰
a. DNA甲基化
DNA甲基化发生在CpG的两个核苷酸上,由DNA甲基转移酶家族的蛋白催化:DNMT1,DNMT3A,DNMT3B和DNMT3L。DNA甲基化会影响DNA构想的变化,削弱DNA与蛋白质之间的相互作用,比如转录因子结合、并激活DNA转录等,从而影响相应位置基因的转录。DNA甲基化能够调控胚胎发育、精子形成、个体发育等等几乎所有的发育过程
(
5
)
。
b. TET蛋白催化DNA去甲基化:
DNA甲基化是一个可逆的过程,能够被两种机制去除:细胞分裂时DNA复制导致丢失一半;也可能由去甲基化酶直接催化,即被甲基胞嘧啶双加氧酶TET家族蛋白(TET1/2/3)催化,形成5-羟甲基胞嘧啶(5hmC)。5hmC通常在位于基因增强子部位。着床前胚胎中的DNA甲基化水平达到最低,在着床后的外胚层中迅速上升。在着床前的早期胚胎发育过程中以及PGC过程中,亲本DNA去甲基化并不依赖于TETs蛋白的功能,而是更多的是随着DNA复制、细胞分裂被动去除;着床后的胚胎发育过程中,DNA去甲基化过程需要TETs去甲基化酶活性
(
5-6
)
。
2 染色体三维结构
分裂间期的基因组按照染色体功能的拓扑结构(topologically associated domains,TADs)分布(Fig.2),这种结构的改变也会对于染色质功能、基因转录和抑制等起到作用。有趣的是TADs在不同细胞类型、分化水平甚至不同物种中间都是相对稳定的。TADs的边界处一般会结合CTCF(76%),可能在限制表观遗传标记的跨TADs功能上起作用。CTCT结合位点去除会导致激活型的染色体修饰错误的传递到周边预定沉默区域,证明TAD作为一个结构性框架能够影响表观遗传信息。多个TADs聚合后有道染色体两种结构形式(A和B)(Fig.2b)。其中A模式下的染色体处于激活状态,而B则是失活状态,且不同细胞分化状态和表观遗传标记下,A/B两种状态可以相互转换
(















